Szerkesztő:Hollófernyiges/próbalap

A genetika vagy örökléstan az élőlények tulajdonságainak öröklődését, a tulajdonságokat kialakító géneket tanulmányozó tudományág. A biológia egyik alapvető ága, mivel az öröklődés ismerete létfontosságú az élőlények evolúciójának megértéséhez. Alapvető törvényszerűségeit Gregor Mendel brünni szerzetes ismerte fel először a 19. században; megfigyelte, hogy a szervezetek (borsónövények) tulajdonságaikat különálló "öröklődési egységek" (ma géneknek nevezzük) révén adják az utódaiknak.
A tulajdonságok öröklődése és a gének molekuláris öröklődési mechanizmusainak megértése továbbra is a genetika elsődleges feladatai, de a 20. század közepétől teendői kibővültek a gének működésének és viselkedésének tanulmányozásával. A tudományág számos részterületre bomlott, ide tartozik többek között a molekuláris genetika, az epigenetika vagy a populációgenetika. A genetika az élővilág valamennyi csoportját (beleértve az eukariótákt, prokariótákat, sőt a nem mindig élőnek tekintett vírusokat is) tanulmányozza.
A genetikai folyamatok az élőlény környezetével együttesen befolyásolják annak tulandonságait vagy viselkedését. A gének kifejeződését a sejten belüli vagy kívüli környezeti tényezők egyaránt növelhetik vagy visszafoghatják.
Története[szerkesztés]
A genetika szó az ógörög γενετικός (genetikosz, jelentése nemző, létrehozó) kifejezésből ered, amely a "származás" jelentésű γένεσις-ből (geneszisz) származtatott.[1][2][3]
Ősidők óta ismert, hogy az utódok öröklik szüleik tulajdonságait. A jelenséget a mezőgazdaságban az állatok és növények háziasítása, nemesítése során széleskörűen felhasználták.[4][5] A modern, tudományos alapokon nyugvó genetika a 19. század közepén, a morvaországi Gregor Mendel ágostonos szerzetes munkásságával kezdődött.[6]
Mendelt megelőzően a magyar Festetics Imre már felismerte az öröklődés egyes törvényszerűségeit és ő használta mai értelemben először a genetika kifejezést. 1819-ben közzétett, juhnemesítési tapasztalait összegző művében A természet genetikai törvényei-ben (Die genetischen Gesetze der Natur) ő is megfogalmazza Mendel második törvényét a nagyszülői tulajdonságok újra felbukkanásáról, valamint a mutációk megjelenéséről is számot ad.[7][8][9]

A 19. században úgy vélték, hogy a szülők tulajdonságai az utódban elkeverednek, átlagolódnak (Darwin is ezt a modellt követte A fajok eredeté-ben).[10] A gyakorlati tapasztalat valóban ezt mutatja számos esetben (pl. testmagasság, bőrszín, stb.), Mendel a munkájában azonban számos olyan példát mutatott, ahol a tulajdonságok egyáltalán nem keveredtek és a következő nemzedékekben teljes mivoltukban újra feltűntek. A látszólagos vegyülés csak a sokgénes tulajdonságoknál lép fel. A másik népszerű öröklődésmodell Jean-Baptiste Lamarck nevéhez fűződik; ő azt állította, hogy a szerzett tulajdonságok is öröklődhetnek, az izmos kovács gyerekei izmosabbak lesznek az átlagnál, a lombért nyújtózkodó zsiráf utódainak hosszabb lesz a nyaka. Elméletét August Weismann kísérletekkel cáfolta.[11] Darwin és követője, Francis Galton is kidolgozta a maga pangenetikus elméletét, miszerint a testrészek és szervek apró, gemmuláknak nevezett részecskéket bocsátanak ki, amelyek az ivarsejtekben koncentrálódnak és így kerülnek át a tulajdonságok a kö vetkező nemzedékbe.[12]
Mendeli genetika[szerkesztés]

A modern genetika Gregor Mendel munkájával kezdődött. 1865-ben "Növényhibridekkel végzett kísérletek" (Versuche über Pflanzenhybriden) címmel bemutatta a brünni Természettudományos Társaságnak (Naturforschender Verein) addigi kutatásainak összegzését. Mendel a borsó bizonyos tulajdonságainak öröklődését vizsgálta nemzedékeken keresztül és eredményeit matematikai módszerekkel is feldolgozta. Bár az általa felfedezett törvényszerűségek nem minden minden tulajdonság esetében voltak megfigyelhetők, de azt bebizonyította, hogy az tulajdonságok különálló "csomagokban" adódnak át és nem keverednek és átadódásuk egyszerű matematikai képletekkel leírható.[13]
A tudományos közvélemény nem ismerte fel azonnal Mendel eredményeinek jelentőségét, csak halála után ismerték el, amikor 1900-ban Hugo de Vries és más kutatók újrafelfedezték törvényeit. A genetika kifejezés is ekkor terjedt el, miután William Bateson 1905-ben javasolta használatát.[14][15]
A Mendel-törvények újrafelfedezését követően a kutatók azt próbálták kideríteni, hogy a sejt mely molekulái felelősek az öröklődését. Az amerikai Nettie Stevens 1905-ben felfedezte, hogy a lisztbogarak nőstényeinek csak X, míg a hímeknek X és Y kromoszómái vannak. Megállapította, hogy a nemet a kromoszómák határozzák meg és attól függ, hogy a hím milyen spermiuma termékenyíti meg a petesejtet.[16] 1911-ben Thomas Hunt Morgan kijelentette, hogy a gének a kromoszómákon kell hogy legyenek, ugyanis megfigyelte, hogy a muslicák fehérszem-mutációja nemhez kötött, azzal együtt öröklődik.[17] Tanítványa, Alfred Sturtevant 1913-ban a génkapcsoltság tanulmányozásával bebizonyította, hogy a gének sorban helyezkednek el a kromoszómán.[18]
Molekuláris genetika[szerkesztés]
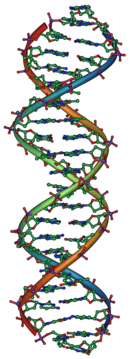
Bár azt már tudták, hogy a gének a kromoszómákon találhatóak, rejtély maradt hogy azok két komponense, a fehérjék és a nukleinsavak közül melyik felelős az öröklődésért. 1928-ban a brit Frederick Griffith felfedezte a transzformáció jelenségét: elpusztult baktériumokból genetikai anyagot átvive élőkre, az utóbbiak megkaptak az előbbiek tulajdonságait, átváltoztak ("transzformálódtak"). Tizenhat évvel később, 1944-ben Oswald Avery bebizonyította, hogy a transzformációt a DNS okozza.[19] 1943-ban Joachim Hämmerling egysejtű algakísérleteivel kimutatta, hogy az eukariótákban a genetikai anyag a sejtmagban található.[20] 1952 Alfred Hershey és Martha Chase bakteriofágokkal dolgozva megerősítette, hogy a vírusok esetében is a DNS (nem pedig a fehérje) az örökletes tulajdonságok hordozója.[21]
1953-ban James Watson és Francis Crick röntgenkrisztallográfiás felvételek segítségével meghatározta a DNS-molekula szerkezetét: kiderült, hogy az két, spirálisan egymás köré tekeredő, rendkívül hosszú szálból áll.[22][23] A polimerszálak alapegységei, a négyféle nukleotid befelé nézve, egymást kiegészítve kapcsolódtak, mint egyfajta megcsavart létra fokai.[24] A szerkezetből magától értetődően következett, hogy a genetikai információ a nukleotidok sorrendjében (szekvenciájában) rejlik. Egyúttal azt is megmagyarázta, hogy hogyan kettőződik meg a DNS a sejtosztódás során; a széttekeredő szálak saját maguk szolgáltak mintául (templátul) a kiegészítő szálnak. A leányDNS-ek egyik szála mindig a régi DNS-ből származik, míg a másik újonnan szintetizálódik (a DNS szemikonzervatív).[25]
Bár a DNS szerkezete magyarázta az öröklődés általános működését, továbbra is rejtély volt, hogy milyen módon befolyásolja a sejt működését. Az elkövetkező években a kutatás elsősorban arra irányult, hogy hogyan szabályozza a DNS a proteinek termelését.[26] Felfedezték, hogy a DNS egyik száláról RNS (ribonukleinsav, ami kémiailag nagyon hasonlít a DNS-re) másolat készül, az ún. hírvivő RNS (mRNS). Ennek nukleotidsorrendje a genetikai kódnak megfelelően fordítódik le a sejt citoplazmájában a fehérje aminosavszekvenciájára.[27]
Az öröklődés és sejtműködés alapelveinek tisztázása számtalan új felfedezés felé nyitotta meg az utat.[28] 1968-ban a japán Kimura Otó megfogalmazta a molekuláris evolúció semleges elméletét, miszerint az evolúció DNS-szinten a semleges (aminosavszekvenciában nem mutatkozó) mutációk felhalmozódásával is folyhat.[29] 1977-ben Frederick Sanger kifejlesztette a technikát a DNS nukleotidsorrendjének leolvasására (szekvenálására).[30] 1983-ban Kary Mullis felfedezett egy módszert, a polimeráz-láncreakciót a DNS egyes szakaszainak felszaporítására, lehetővé téve ezzel a specifikus részek gyors detektálását.[31] 2003-ra számos kutatócsoport nemzetközi együttműködésével (illetve velük párhuzamosan a Celera Genomics magáncég révén) sikeresen befejeződött a Human Genome Project, meghatározták az ember DNS-ének teljes bázissorrendjét.[32][33]
Features of inheritance[szerkesztés]
Discrete inheritance and Mendel's laws[szerkesztés]
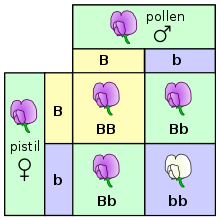
Egyszerűen megfogalmazva az öröklődés az élőlényekben úgy történik, hogy a szülőkről az utódokra különálló öröklési egységek, úgynevezett gének szállnak át.[34] Ezt a jelenséget először Gregor Mendel figyelte meg, aki borsónövényeken tanulmányozta az öröklődő tulajdonságok szétválását, és többek között kimutatta, hogy egy növény virágai vagy lilák vagy fehérek, de soha nem köztes színűek. Ugyanannak a génnek az adott megfigyelhető tulajdonságot (fenotípust) irányító különálló változatait alléloknak nevezzük.[13][35]
A borsó esetében (amely egy diploid faj) a növény minden egyes génből két példányt tartalmaz, véletlenszerűen egy-egy példányt örökölt mindkét szülőjétől.[36] Az állatok és növények többsége (köztük az ember is) hasonlóan működik. Ez a Mendel-törvények között a szétválás szabálya, az allélok egymástól elválva kerülnek az ivarsejtekbe. Azt az élőlényt, amely egy adott gén esetében ugyanazt az allélt örökölte mind a két szülőtől, homozigótának nevezzük (erre a génre vonatkoztatva), míg azokat amelyekben két különböző allél található, heterozigótának hívjuk. Az élőlény teljes génkészletének neve genotípus, míg a megfigyelhető tulajdonságaié fenotípus. A heterozigótáknál sok esetben csak az egyik allél hatása figyelhető meg a fenotípusban, a dominás allél elnyomja a recesszívet (a fenti példában a virág lilaságát meghatározó allél elnyomja a fehér szín allélját). Máskor a dominancia nem teljes és köztes fenotípus mutatkozik meg vagy kodomináns állapot lép fel, amikor mindkét allél egyformán kifejeződik.[37]
A domináns és recesszív allélok miatt azonban a fenotípus eltérhez a genotípustól. Mendel kísérleteiben kiderült, hogy a virágszínt vizsgálva az első nemzedék teljes egészében lila lett, míg ha ezeket egymás között tovább keresztezte, a második generáció növényeiben 3:1 arányban ismét megjelent a fehér szín.
Multiple gene interactions[szerkesztés]

Az élőlényeknek több ezer vagy tízezer génjük van, és az ivarosan szaporodó szervezetekben ezek a gének általában egymástól függetlenül rendeződnek; vagyis a borsószem sárga vagy zöld színét meghatározó allél öröklődése nincs kapcsolatban a fehér vagy lila virágok alléljának öröklődésével. Ezt a jelenséget Mendel második törvényének vagy a "független öröklődés törvényének" nevezik. Ennek hatására a gének alléljai az utódokban különféle kombinációkat hozhatnak létre. A gének sokszor egymással kölcsönhatva befolyásolják ugyanazt a tulajdonságot. A tavaszi békaszemnek (Omphalodes verna) például van egy génje, amelynek alléljai meghatározzák a virágok színét: kék vagy lila lehet. Egy másik gén viszont azt szabályozza, hogy a virágok egyáltalán színesek vagy inkább pigment nélküli fehérek legyenek. Ha egy növénynek két példánya van a fehér allélből, akkor a virágai fehérek lesznek, függetlenül attól, hogy az első gén kék vagy lila alléllal rendelkezik-e. Az efféle gének közötti kölcsönhatást episztázisnak nevezzük, a második gén episztatikusan hat az elsőre.[38]
Sok tulajdonság nem "bináris" jellegű (pl. vagy lila vagy fehér virág), hanem értéke folytonos skála mentén változik (ilyen pl. az ember magassága vagy bőrszíne). Ezeket általában sok gén befolyásolja egyszerre, ún. multigénes tulajdonságok.[39] A helyzetet tovább bonyolítja, hogy a végső tulajdonságra a környezet is jelentős hatást gyakorolhat (pl. gyerekkori éhezés a testmagasságra). Azt, hogy egy élőlény génjei mekkora mértékben járulnak hozzá egy tulajdonsághoz, heritabilitásnak nevezzük.[40] A heritabilitás értéke is különböző külső hatásoktól függhet, pl. a testmagasságnak az Egyesült Államokban 89%-os a heritabilitása, míg Nigériában (ahol a gyerekek élelmiszerhez és egészségügyi ellátáshoz való hozzáférése sokkal inkább függ a társadalmi helyzettől) mindössze 62%.[41]
Az öröklődés molekuláris alapjai[szerkesztés]
A DNS és a kromoszómák[szerkesztés]

A gének molekuláris szempontból a dezoxiribonukleinsavban (röviden DNS) testesülnek meg. A DNS egy hosszú lánc, amely egymással váltakozó dezoxiribóz (egyfajta cukormolekula) és foszfát molekuláiból áll és a cukorhoz oldalvást nitrogéntartalmú bázisok kapcsolódnak. Négy ilyen bázis létezik: az adenin (A), citozin (C), guanin (G) és a timin (T). A láncok oldalsó bázisai hidrogénkötésekkel egymáshoz kapcsolódnak (az adenin csak timinnel, a guanin csak citozinnal), hosszú kétszálú, létraszerű struktúrát hozva létre, amely spirálisan csavarodik: ez a híres kettős spirál.[42] A genetikai információ a bázisok sorrendjében (szekvenciájában) rejlik, a gének gyakorlatilag a hosszú DNS-lánc egyes szakaszainak felelnek meg.[43] A sejtben a rendkívül hosszú DNS-molekulák hisztonfehérjékre feltekeredve helyezkednek el, hogy kevesebb helyet foglaljanak. Az így felcsavart egyetlen DNS-molekula a kromoszóma.[44] Egyes vírusok DNS helyett a kémiailag igen hasonló ribonukleinsavat (RNS) használják genetikai információik tárolására.[45]
Mivel a kettős spirál szálain a szemben levő bázisok kiegészítik egymást, mindkét szál külön-külön is magában hordozza a teljes genetikai információt. A DNS másolásakor a szálak szétválnak és mindkettő mintaként (templátként) szolgál az új kiegészítő szál szintéziséhez.[46]

A gének lineárisan, egymást követően helyezkednek el a DNS hosszú szálain. A baktériumok génjei jellemzően egyetlen, kör alakú DNS-en találhatók, míg az eukarióták (mint az állatok és növények) sejtjei több, különböző hosszúságú DNS-láncon (kromoszómán) tárolják génjeiket. A DNS-szálak sokszor rendkívül hosszúak; például a legnagyobb emberi kromoszóma (az I-es) 247 millió bázispár hosszú.[47] A kromoszóma DNS-éhez fehérjék kapcsolódnak, amelyek segítenek kezelhető méretűre csomagolni azt és szabályozni az egyes génszakaszok hozzáférhetőségét, átírhatóságát. Azt a DNS-fehérje komplexet kromatinnak hívják; az eukarióták esetében ennek alapegysége a nukleoszóma, a hisztonfehérjék köré tekert DNS.[48] A szervezet teljes genetikai információjának összességét (általában kromoszómáinak összesített szekvenciáját) genomnak hívjuk.
A DNS leggyakrabban a sejtmagban található, de egyes sejtszervecskék, mint a mitokondriumok vagy a növények kloroplasztiszai is tartalmaznak valamennyit belőle.
A legtöbb állat és növény minden kromoszómából két példányt tartalmaz, vagyis diploidok (léteznek egy kromoszómakészlettel rendelkező, ún. haploid eukarióták is, mint pl. a mohák). Az egyik példány az anyától, a másik az apától származik.[36]
Sok faj rendelkezik nemi kromoszómával, amelyek megléte vagy hiánya határozza meg az egyed nemét.[49] Az emlősök (és így az ember) esetében a nőstényeknek két egyforma nemi kromoszómájuk (XX) van, míg a hímek XY készlettel rendelkeznek; az Y kromoszómán találhatók meg azok a gének, amelyek elidítják a rájuk jellemző tulajdonságok kifejlődését. Az evolúció során az Y kromoszóma elvesztette génjei többségét és jóval kisebb lett az X-nél, amely átlagos méretű. Emiatt a nőstények a rajta található génekből két példánnyal rendelkeznek, míg a hímek csak eggyel. Hogy az ezzel járó biokémiai problémákat kiküszöbölják, a nőstények sejtjeiben az egyik, véletéenszerűen kiválasztott X kromoszóma kikapcsolódik.[50] Mivel a hímek az X kromoszóma génjeire haploidok, ezért náluk jóval gyakrabban manifesztálódnak az ezekhez kapcsolódó genetikai betegségek is.[50] Más élőlények nemmeghatározó rendszere másképp működik, pl. a madaraknál a hímeknek vannak egyforma nemi kromoszómáik (ZZ), a tojóké pedig különböző (ZW).
A szaporodás[szerkesztés]

When cells divide, their full genome is copied and each daughter cell inherits one copy. This process, called mitosis, is the simplest form of reproduction and is the basis for asexual reproduction. Asexual reproduction can also occur in multicellular organisms, producing offspring that inherit their genome from a single parent. Offspring that are genetically identical to their parents are called clones.[51]
Eukaryotic organisms often use sexual reproduction to generate offspring that contain a mixture of genetic material inherited from two different parents. The process of sexual reproduction alternates between forms that contain single copies of the genome (haploid) and double copies (diploid).[36] Haploid cells fuse and combine genetic material to create a diploid cell with paired chromosomes. Diploid organisms form haploids by dividing, without replicating their DNA, to create daughter cells that randomly inherit one of each pair of chromosomes. Most animals and many plants are diploid for most of their lifespan, with the haploid form reduced to single cell gametes such as sperm or eggs.[52]
Although they do not use the haploid/diploid method of sexual reproduction, bacteria have many methods of acquiring new genetic information. Some bacteria can undergo conjugation, transferring a small circular piece of DNA to another bacterium.[53] Bacteria can also take up raw DNA fragments found in the environment and integrate them into their genomes, a phenomenon known as transformation.[54] These processes result in horizontal gene transfer, transmitting fragments of genetic information between organisms that would be otherwise unrelated. Natural bacterial transformation occurs in many bacterial species, and can be regarded as a sexual process for transferring DNA from one cell to another cell (usually of the same species).[55] Transformation requires the action of numerous bacterial gene products, and its primary adaptive function appears to be repair of DNA damages in the recipient cell.[55]
Recombination and genetic linkage[szerkesztés]

The diploid nature of chromosomes allows for genes on different chromosomes to assort independently or be separated from their homologous pair during sexual reproduction wherein haploid gametes are formed. In this way new combinations of genes can occur in the offspring of a mating pair. Genes on the same chromosome would theoretically never recombine. However, they do, via the cellular process of chromosomal crossover. During crossover, chromosomes exchange stretches of DNA, effectively shuffling the gene alleles between the chromosomes.[56] This process of chromosomal crossover generally occurs during meiosis, a series of cell divisions that creates haploid cells. Meiotic recombination, particularly in microbial eukaryotes, appears to serve the adaptive function of repair of DNA damages.[55]
The first cytological demonstration of crossing over was performed by Harriet Creighton and Barbara McClintock in 1931. Their research and experiments on corn provided cytological evidence for the genetic theory that linked genes on paired chromosomes do in fact exchange places from one homolog to the other.[57]
The probability of chromosomal crossover occurring between two given points on the chromosome is related to the distance between the points. For an arbitrarily long distance, the probability of crossover is high enough that the inheritance of the genes is effectively uncorrelated.[58] For genes that are closer together, however, the lower probability of crossover means that the genes demonstrate genetic linkage; alleles for the two genes tend to be inherited together. The amounts of linkage between a series of genes can be combined to form a linear linkage map that roughly describes the arrangement of the genes along the chromosome.[59]
Gene expression[szerkesztés]
Genetic code[szerkesztés]
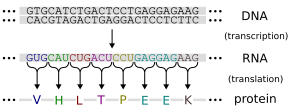
Genes express their functional effect through the production of proteins, which are molecules responsible for most functions in the cell. Proteins are made up of one or more polypeptide chains, each composed of a sequence of amino acids. The DNA sequence of a gene is used to produce a specific amino acid sequence. This process begins with the production of an RNA molecule with a sequence matching the gene's DNA sequence, a process called transcription.
This messenger RNA molecule then serves to produce a corresponding amino acid sequence through a process called translation. Each group of three nucleotides in the sequence, called a codon, corresponds either to one of the twenty possible amino acids in a protein or an instruction to end the amino acid sequence; this correspondence is called the genetic code.[60] The flow of information is unidirectional: information is transferred from nucleotide sequences into the amino acid sequence of proteins, but it never transfers from protein back into the sequence of DNA—a phenomenon Francis Crick called the central dogma of molecular biology.[61]
The specific sequence of amino acids results in a unique three-dimensional structure for that protein, and the three-dimensional structures of proteins are related to their functions.[62][63] Some are simple structural molecules, like the fibers formed by the protein collagen. Proteins can bind to other proteins and simple molecules, sometimes acting as enzymes by facilitating chemical reactions within the bound molecules (without changing the structure of the protein itself). Protein structure is dynamic; the protein hemoglobin bends into slightly different forms as it facilitates the capture, transport, and release of oxygen molecules within mammalian blood.[forrás?]
A single nucleotide difference within DNA can cause a change in the amino acid sequence of a protein. Because protein structures are the result of their amino acid sequences, some changes can dramatically change the properties of a protein by destabilizing the structure or changing the surface of the protein in a way that changes its interaction with other proteins and molecules. For example, sickle-cell anemia is a human genetic disease that results from a single base difference within the coding region for the β-globin section of hemoglobin, causing a single amino acid change that changes hemoglobin's physical properties.[64] Sickle-cell versions of hemoglobin stick to themselves, stacking to form fibers that distort the shape of red blood cells carrying the protein. These sickle-shaped cells no longer flow smoothly through blood vessels, having a tendency to clog or degrade, causing the medical problems associated with this disease.[forrás?]
Some DNA sequences are transcribed into RNA but are not translated into protein products—such RNA molecules are called non-coding RNA. In some cases, these products fold into structures which are involved in critical cell functions (e.g. ribosomal RNA and transfer RNA). RNA can also have regulatory effects through hybridization interactions with other RNA molecules (such as microRNA).[forrás?]
Nature and nurture[szerkesztés]

Although genes contain all the information an organism uses to function, the environment plays an important role in determining the ultimate phenotypes an organism displays. The phrase "nature and nurture" refers to this complementary relationship. The phenotype of an organism depends on the interaction of genes and the environment. An interesting example is the coat coloration of the Siamese cat. In this case, the body temperature of the cat plays the role of the environment. The cat's genes code for dark hair, thus the hair-producing cells in the cat make cellular proteins resulting in dark hair. But these dark hair-producing proteins are sensitive to temperature (i.e. have a mutation causing temperature-sensitivity) and denature in higher-temperature environments, failing to produce dark-hair pigment in areas where the cat has a higher body temperature. In a low-temperature environment, however, the protein's structure is stable and produces dark-hair pigment normally. The protein remains functional in areas of skin that are colder—such as its legs, ears, tail, and faceSablon:Emdashso the cat has dark hair at its extremities.[65]
Environment plays a major role in effects of the human genetic disease phenylketonuria. The mutation that causes phenylketonuria disrupts the ability of the body to break down the amino acid phenylalanine, causing a toxic build-up of an intermediate molecule that, in turn, causes severe symptoms of progressive intellectual disability and seizures. However, if someone with the phenylketonuria mutation follows a strict diet that avoids this amino acid, they remain normal and healthy.[66]
A common method for determining how genes and environment ("nature and nurture") contribute to a phenotype involves studying identical and fraternal twins, or other siblings of multiple births.[67] Identical siblings are genetically the same since they come from the same zygote. Meanwhile, fraternal twins are as genetically different from one another as normal siblings. By comparing how often a certain disorder occurs in a pair of identical twins to how often it occurs in a pair of fraternal twins, scientists can determine whether that disorder is caused by genetic or postnatal environmental factors. One famous example involved the study of the Genain quadruplets, who were identical quadruplets all diagnosed with schizophrenia.[68]
Gene regulation[szerkesztés]
The genome of a given organism contains thousands of genes, but not all these genes need to be active at any given moment. A gene is expressed when it is being transcribed into mRNA and there exist many cellular methods of controlling the expression of genes such that proteins are produced only when needed by the cell. Transcription factors are regulatory proteins that bind to DNA, either promoting or inhibiting the transcription of a gene.[69] Within the genome of Escherichia coli bacteria, for example, there exists a series of genes necessary for the synthesis of the amino acid tryptophan. However, when tryptophan is already available to the cell, these genes for tryptophan synthesis are no longer needed. The presence of tryptophan directly affects the activity of the genes—tryptophan molecules bind to the tryptophan repressor (a transcription factor), changing the repressor's structure such that the repressor binds to the genes. The tryptophan repressor blocks the transcription and expression of the genes, thereby creating negative feedback regulation of the tryptophan synthesis process.[70]
Differences in gene expression are especially clear within multicellular organisms, where cells all contain the same genome but have very different structures and behaviors due to the expression of different sets of genes. All the cells in a multicellular organism derive from a single cell, differentiating into variant cell types in response to external and intercellular signals and gradually establishing different patterns of gene expression to create different behaviors. As no single gene is responsible for the development of structures within multicellular organisms, these patterns arise from the complex interactions between many cells.[forrás?]
Within eukaryotes, there exist structural features of chromatin that influence the transcription of genes, often in the form of modifications to DNA and chromatin that are stably inherited by daughter cells.[71] These features are called "epigenetic" because they exist "on top" of the DNA sequence and retain inheritance from one cell generation to the next. Because of epigenetic features, different cell types grown within the same medium can retain very different properties. Although epigenetic features are generally dynamic over the course of development, some, like the phenomenon of paramutation, have multigenerational inheritance and exist as rare exceptions to the general rule of DNA as the basis for inheritance.[72]
Genetic change[szerkesztés]
Mutations[szerkesztés]

During the process of DNA replication, errors occasionally occur in the polymerization of the second strand. These errors, called mutations, can affect the phenotype of an organism, especially if they occur within the protein coding sequence of a gene. Error rates are usually very low—1 error in every 10–100 million bases—due to the "proofreading" ability of DNA polymerases.[73][74] Processes that increase the rate of changes in DNA are called mutagenic: mutagenic chemicals promote errors in DNA replication, often by interfering with the structure of base-pairing, while UV radiation induces mutations by causing damage to the DNA structure.[75] Chemical damage to DNA occurs naturally as well and cells use DNA repair mechanisms to repair mismatches and breaks. The repair does not, however, always restore the original sequence. A particularly important source of DNA damages appears to be reactive oxygen species[76] produced by cellular aerobic respiration, and these can lead to mutations.[77]
In organisms that use chromosomal crossover to exchange DNA and recombine genes, errors in alignment during meiosis can also cause mutations. Errors in crossover are especially likely when similar sequences cause partner chromosomes to adopt a mistaken alignment; this makes some regions in genomes more prone to mutating in this way. These errors create large structural changes in DNA sequence—duplications, inversions, deletions of entire regions—or the accidental exchange of whole parts of sequences between different chromosomes, chromosomal translocation.[78]

Natural selection and evolution[szerkesztés]
Mutations alter an organism's genotype and occasionally this causes different phenotypes to appear. Most mutations have little effect on an organism's phenotype, health, or reproductive fitness.[79] Mutations that do have an effect are usually detrimental, but occasionally some can be beneficial.[80] Studies in the fly Drosophila melanogaster suggest that if a mutation changes a protein produced by a gene, about 70 percent of these mutations are harmful with the remainder being either neutral or weakly beneficial.[81]

Population genetics studies the distribution of genetic differences within populations and how these distributions change over time.[82] Changes in the frequency of an allele in a population are mainly influenced by natural selection, where a given allele provides a selective or reproductive advantage to the organism,[83] as well as other factors such as mutation, genetic drift, genetic hitchhiking,[84] artificial selection and migration.[85]
Over many generations, the genomes of organisms can change significantly, resulting in evolution. In the process called adaptation, selection for beneficial mutations can cause a species to evolve into forms better able to survive in their environment.[86] New species are formed through the process of speciation, often caused by geographical separations that prevent populations from exchanging genes with each other.[87]
By comparing the homology between different species' genomes, it is possible to calculate the evolutionary distance between them and when they may have diverged. Genetic comparisons are generally considered a more accurate method of characterizing the relatedness between species than the comparison of phenotypic characteristics. The evolutionary distances between species can be used to form evolutionary trees; these trees represent the common descent and divergence of species over time, although they do not show the transfer of genetic material between unrelated species (known as horizontal gene transfer and most common in bacteria).[88]
Model organisms[szerkesztés]

Although geneticists originally studied inheritance in a wide variety of organisms, the range of species studied has narrowed. One reason is that when significant research already exists for a given organism, new researchers are more likely to choose it for further study, and so eventually a few model organisms became the basis for most genetics research. Common research topics in model organism genetics include the study of gene regulation and the involvement of genes in development and cancer. Organisms were chosen, in part, for convenience—short generation times and easy genetic manipulation made some organisms popular genetics research tools. Widely used model organisms include the gut bacterium Escherichia coli, the plant Arabidopsis thaliana, baker's yeast (Saccharomyces cerevisiae), the nematode Caenorhabditis elegans, the common fruit fly (Drosophila melanogaster), the zebrafish (Danio rerio), and the common house mouse (Mus musculus).[89]
Medicine[szerkesztés]

Medical genetics seeks to understand how genetic variation relates to human health and disease.[90] When searching for an unknown gene that may be involved in a disease, researchers commonly use genetic linkage and genetic pedigree charts to find the location on the genome associated with the disease. At the population level, researchers take advantage of Mendelian randomization to look for locations in the genome that are associated with diseases, a method especially useful for multigenic traits not clearly defined by a single gene.[91] Once a candidate gene is found, further research is often done on the corresponding (or homologous) genes of model organisms. In addition to studying genetic diseases, the increased availability of genotyping methods has led to the field of pharmacogenetics: the study of how genotype can affect drug responses.[92]
Individuals differ in their inherited tendency to develop cancer, and cancer is a genetic disease. The process of cancer development in the body is a combination of events. Mutations occasionally occur within cells in the body as they divide. Although these mutations will not be inherited by any offspring, they can affect the behavior of cells, sometimes causing them to grow and divide more frequently. There are biological mechanisms that attempt to stop this process; signals are given to inappropriately dividing cells that should trigger cell death, but sometimes additional mutations occur that cause cells to ignore these messages. An internal process of natural selection occurs within the body and eventually mutations accumulate within cells to promote their own growth, creating a cancerous tumor that grows and invades various tissues of the body. Normally, a cell divides only in response to signals called growth factors and stops growing once in contact with surrounding cells and in response to growth-inhibitory signals. It usually then divides a limited number of times and dies, staying within the epithelium where it is unable to migrate to other organs. To become a cancer cell, a cell has to accumulate mutations in a number of genes (three to seven). A cancer cell can divide without growth factor and ignores inhibitory signals. Also, it is immortal and can grow indefinitely, even after it makes contact with neighboring cells. It may escape from the epithelium and ultimately from the primary tumor. Then, the escaped cell can cross the endothelium of a blood vessel and get transported by the bloodstream to colonize a new organ, forming deadly metastasis. Although there are some genetic predispositions in a small fraction of cancers, the major fraction is due to a set of new genetic mutations that originally appear and accumulate in one or a small number of cells that will divide to form the tumor and are not transmitted to the progeny (somatic mutations). The most frequent mutations are a loss of function of p53 protein, a tumor suppressor, or in the p53 pathway, and gain of function mutations in the Ras proteins, or in other oncogenes.[93][94]
Research methods[szerkesztés]
DNA can be manipulated in the laboratory. Restriction enzymes are commonly used enzymes that cut DNA at specific sequences, producing predictable fragments of DNA.[95] DNA fragments can be visualized through use of gel electrophoresis, which separates fragments according to their length.[forrás?]
The use of ligation enzymes allows DNA fragments to be connected. By binding ("ligating") fragments of DNA together from different sources, researchers can create recombinant DNA, the DNA often associated with genetically modified organisms. Recombinant DNA is commonly used in the context of plasmids: short circular DNA molecules with a few genes on them. In the process known as molecular cloning, researchers can amplify the DNA fragments by inserting plasmids into bacteria and then culturing them on plates of agar (to isolate clones of bacteria cells). "Cloning" can also refer to the various means of creating cloned ("clonal") organisms.[96]
DNA can also be amplified using a procedure called the polymerase chain reaction (PCR).[97] By using specific short sequences of DNA, PCR can isolate and exponentially amplify a targeted region of DNA. Because it can amplify from extremely small amounts of DNA, PCR is also often used to detect the presence of specific DNA sequences.[forrás?]
DNA sequencing and genomics[szerkesztés]
DNA sequencing, one of the most fundamental technologies developed to study genetics, allows researchers to determine the sequence of nucleotides in DNA fragments. The technique of chain-termination sequencing, developed in 1977 by a team led by Frederick Sanger, is still routinely used to sequence DNA fragments. Using this technology, researchers have been able to study the molecular sequences associated with many human diseases.[98]
As sequencing has become less expensive, researchers have sequenced the genomes of many organisms using a process called genome assembly, which uses computational tools to stitch together sequences from many different fragments.[99] These technologies were used to sequence the human genome in the Human Genome Project completed in 2003.[32] New high-throughput sequencing technologies are dramatically lowering the cost of DNA sequencing, with many researchers hoping to bring the cost of resequencing a human genome down to a thousand dollars.[100]
Next-generation sequencing (or high-throughput sequencing) came about due to the ever-increasing demand for low-cost sequencing. These sequencing technologies allow the production of potentially millions of sequences concurrently.[101][102] The large amount of sequence data available has created the subfield of genomics, research that uses computational tools to search for and analyze patterns in the full genomes of organisms. Genomics can also be considered a subfield of bioinformatics, which uses computational approaches to analyze large sets of biological data. A common problem to these fields of research is how to manage and share data that deals with human subject and personally identifiable information.[forrás?]
Society and culture[szerkesztés]
On 19 March 2015, a group of leading biologists urged a worldwide ban on clinical use of methods, particularly the use of CRISPR and zinc finger, to edit the human genome in a way that can be inherited.[103][104][105][106] In April 2015, Chinese researchers reported results of basic research to edit the DNA of non-viable human embryos using CRISPR.[107][108]
See also[szerkesztés]
- Bacterial genome size
- Cryoconservation of animal genetic resources
- Eugenics
- Embryology
- Genetic disorder
- Genetic diversity
- Genetic engineering
- Genetic enhancement
- Glossary of genetics (M−Z)
- Index of genetics articles
- Medical genetics
- Molecular tools for gene study
- Neuroepigenetics
- Outline of genetics
- Timeline of the history of genetics
- Plant genetic resources
References[szerkesztés]
- ↑ Genetikos (γενετ-ικός). Henry George Liddell, Robert Scott, A Greek-English Lexicon. Perseus Digital Library, Tufts University. [2010. június 15-i dátummal az eredetiből archiválva]. (Hozzáférés: 2012. február 20.)
- ↑ Genesis (γένεσις). Henry George Liddell, Robert Scott, A Greek-English Lexicon. Perseus Digital Library, Tufts University. [2010. június 15-i dátummal az eredetiből archiválva]. (Hozzáférés: 2012. február 20.)
- ↑ Sablon:Cite dictionary
- ↑ Science: The Definitive Visual Guide. Penguin, 362. o. (2009). ISBN 978-0-7566-6490-9
- ↑ (2022. július 1.) „Themes of Biological Inheritance in Early Nineteenth Century Sheep Breeding as Revealed by J. M. Ehrenfels”. Genes 13 (8), 1311. o. DOI:10.3390/genes13081311. PMID 35893050.
- ↑ (1991. július 1.) „Historical study: Johann Gregor Mendel 1822-1884”. American Journal of Medical Genetics 40 (1), 1–25; discussion 26. o. DOI:10.1002/ajmg.1320400103. PMID 1887835.
- ↑ Forráshivatkozás-hiba: Érvénytelen
<ref>címke; nincs megadva szöveg a(z)Poczai and Santiago-Blay 2021nevű lábjegyzeteknek - ↑ (2019. június 1.) „The emergence of genetics from Festetics' sheep through Mendel's peas to Bateson's chickens”. Journal of Genetics 98 (2), 63. o. DOI:10.1007/s12041-019-1108-z. PMID 31204695.
- ↑ (2014. január 1.) „Imre Festetics and the Sheep Breeders' Society of Moravia: Mendel's Forgotten "Research Network"”. PLOS Biology 12 (1), e1001772. o. DOI:10.1371/journal.pbio.1001772. PMID 24465180.
- ↑ Population Genetics. Georgetown University, 26. o. (2011). ISBN 978-1-4443-6245-9
- ↑ Lamarck, J-B (2008). Encyclopædia Britannica. Encyclopædia Britannica Online Archiválva 2020. április 14-i dátummal a Wayback Machine-ben. on 16 March 2008.
- ↑ Peter J. Bowler, The Mendelian Revolution: The Emergency of Hereditarian Concepts in Modern Science and Society (Baltimore: Johns Hopkins University Press, 1989): chapters 2 & 3.
- ↑ a b Mendel's Paper in English. [2016. január 13-i dátummal az eredetiből archiválva].
- ↑ genetics, n., Oxford English Dictionary, 3rd ed.
- ↑ Letter from William Bateson to Alan Sedgwick in 1905. The John Innes Centre. [2007. október 13-i dátummal az eredetiből archiválva]. (Hozzáférés: 2008. március 15.)
- ↑ Forráshivatkozás-hiba: Érvénytelen
<ref>címke; nincs megadva szöveg a(z)netnevű lábjegyzeteknek - ↑ (1983) „Thomas Hunt Morgan – The Geneticist”. Integrative and Comparative Biology 23 (4), 855–865. o. DOI:10.1093/icb/23.4.855.
- ↑ (1913) „The linear arrangement of six sex-linked factors in Drosophila, as shown by their mode of association”. Journal of Experimental Biology 14 (1), 43–59. o. DOI:10.1002/jez.1400140104.
- ↑ (1944. február 1.) „STUDIES ON THE CHEMICAL NATURE OF THE SUBSTANCE INDUCING TRANSFORMATION OF PNEUMOCOCCAL TYPES : INDUCTION OF TRANSFORMATION BY A DESOXYRIBONUCLEIC ACID FRACTION ISOLATED FROM PNEUMOCOCCUS TYPE III”. The Journal of Experimental Medicine 79 (2), 137–158. o. DOI:10.1084/jem.79.2.137. PMID 19871359.
- ↑ Cell and Molecular Biology. I.K. International Pvt Ltd, 221. o. (2008. május 28.). ISBN 978-81-89866-59-4
- ↑ (1952. május 1.) „Independent functions of viral protein and nucleic acid in growth of bacteriophage”. The Journal of General Physiology 36 (1), 39–56. o. DOI:10.1085/jgp.36.1.39. PMID 12981234.
- ↑ The Eighth Day of Creation: Makers of the Revolution in Biology. Cold Spring Harbor Laboratory Press, 51–169. o. (1979). ISBN 978-0-87969-477-7
- ↑ (1953. április 1.) „Molecular structure of nucleic acids; a structure for deoxyribose nucleic acid”. Nature 171 (4356), 737–738. o. DOI:10.1038/171737a0. PMID 13054692.
- ↑ (1953. május 1.) „Genetical implications of the structure of deoxyribonucleic acid”. Nature 171 (4361), 964–967. o. DOI:10.1038/171964b0. PMID 13063483.
- ↑ (2014. február 1.) „DNA replication at the single-molecule level”. Chemical Society Reviews 43 (4), 1201–1220. o. DOI:10.1039/c3cs60391a. PMID 24395040.
- ↑ Managing Science: Methodology and Organization of Research. Springer, 76. o. (2010). ISBN 978-1-4419-7488-4
- ↑ Encyclopedia of Evolution. Infobase Publishing, 134. o. (2009). ISBN 978-1-4381-1005-9
- ↑ Genetics and Reductionism. Cambridge University Press, 140. o. (1998). ISBN 978-0-521-63713-8
- ↑ Ohta T., Gillespie J.H. (1996). „Development of neutral and nearly neutral theories”. Theoretical Population Biology 49 (2), 128–142. o. DOI:10.1006/tpbi.1996.0007. PMID 8813019.
- ↑ (1977. december 1.) „DNA sequencing with chain-terminating inhibitors”. Proceedings of the National Academy of Sciences of the United States of America 74 (12), 5463–5467. o. DOI:10.1073/pnas.74.12.5463. PMID 271968.
- ↑ (1985. december 1.) „Enzymatic amplification of beta-globin genomic sequences and restriction site analysis for diagnosis of sickle cell anemia”. Science 230 (4732), 1350–1354. o. DOI:10.1126/science.2999980. PMID 2999980.
- ↑ a b Human Genome Project Information. Human Genome Project. [2008. március 15-i dátummal az eredetiből archiválva]. (Hozzáférés: 2008. március 15.)
- ↑ „The sequence of the human genome”. Science 291.
- ↑ Patterns of Inheritance: Introduction, An Introduction to Genetic Analysis, 7th, New York: W.H. Freeman (2000). ISBN 978-0-7167-3520-5
- ↑ Mendel's experiments, An Introduction to Genetic Analysis, 7th, New York: W.H. Freeman (2000). ISBN 978-0-7167-3520-5
- ↑ a b c Mendelian genetics in eukaryotic life cycles, An Introduction to Genetic Analysis, 7th, New York: W.H. Freeman (2000). ISBN 978-0-7167-3520-5
- ↑ Interactions between the alleles of one gene, An Introduction to Genetic Analysis, 7th, New York: W.H. Freeman (2000). ISBN 978-0-7167-3520-5
- ↑ Gene interaction and modified dihybrid ratios, An Introduction to Genetic Analysis, 7th, New York: W.H. Freeman (2000). ISBN 978-0-7167-3520-5
- ↑ (2005. június 1.) „Mapping the new frontier: complex genetic disorders”. The Journal of Clinical Investigation 115 (6), 1404–1407. o. DOI:10.1172/JCI25421. PMID 15931374.
- ↑ Quantifying heritability, An Introduction to Genetic Analysis, 7th, New York: W. H. Freeman (2000). ISBN 978-0-7167-3520-5
- ↑ (2001. július 1.) „Heritability of obesity-related traits among Nigerians, Jamaicans and US black people”. International Journal of Obesity and Related Metabolic Disorders 25 (7), 1034–1041. o. DOI:10.1038/sj.ijo.0801650. PMID 11443503.
- ↑ Campbell Biology. plus.pearson.com . (Hozzáférés: 2022. szeptember 28.)
- ↑ (2006. május 1.) „Genetics: what is a gene?”. Nature 441 (7092), 398–401. o. DOI:10.1038/441398a. PMID 16724031.
- ↑ Histone (angol nyelven). Genome.gov . (Hozzáférés: 2022. szeptember 28.)
- ↑ Microbiology, 3rd, Wm. C. Brown, 343. o. (1996). ISBN 0-697-21865-1
- ↑ Mechanism of DNA Replication, An Introduction to Genetic Analysis, 7th, New York: W.H. Freeman (2000). ISBN 978-0-7167-3520-5
- ↑ (2006. május 1.) „The DNA sequence and biological annotation of human chromosome 1”. Nature 441 (7091), 315–321. o. DOI:10.1038/nature04727. PMID 16710414.
- ↑ Alberts et al. (2002), II.4. DNA and chromosomes: Chromosomal DNA and Its Packaging in the Chromatin Fiber Archiválva 2007. október 18-i dátummal a Wayback Machine-ben.
- ↑ Sex chromosomes and sex-linked inheritance, An Introduction to Genetic Analysis, 7th, New York: W.H. Freeman (2000). ISBN 978-0-7167-3520-5
- ↑ a b (2015. február 1.) „Mary F. Lyon (1925-2014)”. Nature 518 (7537), 36. o, Kiadó: Springer Nature Limited. DOI:10.1038/518036a. PMID 25652989.
- ↑ clone. Merriam-Webster Dictionary . (Hozzáférés: 2023. november 13.)
- ↑ Haploid (angol nyelven). www.genome.gov . (Hozzáférés: 2024. február 10.)
- ↑ Bacterial conjugation, An Introduction to Genetic Analysis, 7th, New York: W.H. Freeman (2000). ISBN 978-0-7167-3520-5
- ↑ Bacterial transformation, An Introduction to Genetic Analysis, 7th, New York: W.H. Freeman (2000). ISBN 978-0-7167-3520-5
- ↑ a b c (2018. január 1.) „Sex in microbial pathogens”. Infection, Genetics and Evolution 57, 8–25. o. DOI:10.1016/j.meegid.2017.10.024. PMID 29111273.
- ↑ Nature of crossing-over, An Introduction to Genetic Analysis, 7th, New York: W. H. Freeman (2000). ISBN 978-0-7167-3520-5
- ↑ (1931. augusztus 1.) „A Correlation of Cytological and Genetical Crossing-Over in Zea Mays”. Proceedings of the National Academy of Sciences of the United States of America 17 (8), 492–497. o. DOI:10.1073/pnas.17.8.492. PMID 16587654.
- ↑ Crossover: Concepts and Applications in Genetics, Evolution, and Breeding. University of Wisconsin Press, 55. o. (1994). ISBN 978-0-299-13564-5
- ↑ Linkage maps, An Introduction to Genetic Analysis, 7th, New York: W. H. Freeman (2000). ISBN 978-0-7167-3520-5
- ↑ I. 5. DNA, RNA, and the Flow of Genetic Information: Amino Acids Are Encoded by Groups of Three Bases Starting from a Fixed Point, Biochemistry, 5th, New York: W.H. Freeman and Company (2002)
- ↑ (1970. augusztus 1.) „Central dogma of molecular biology”. Nature 227 (5258), 561–563. o. DOI:10.1038/227561a0. PMID 4913914.
- ↑ Alberts et al. (2002), I.3. Proteins: The Shape and Structure of Proteins Archiválva 2023. január 1-ji dátummal a Wayback Machine-ben.
- ↑ Alberts et al. (2002), I.3. Proteins: Protein Function Archiválva 2006. április 25-i dátummal a Wayback Machine-ben.
- ↑ How Does Sickle Cell Cause Disease?. Brigham and Women's Hospital: Information Center for Sickle Cell and Thalassemic Disorders, 2002. április 11. [2010. szeptember 23-i dátummal az eredetiből archiválva]. (Hozzáférés: 2007. július 23.)
- ↑ (2006. április 1.) „Albinism in the domestic cat (Felis catus) is associated with a tyrosinase (TYR) mutation”. Animal Genetics 37 (2), 175–178. o. DOI:10.1111/j.1365-2052.2005.01409.x. PMID 16573534.
- ↑ MedlinePlus: Phenylketonuria. NIH: National Library of Medicine. [2008. július 25-i dátummal az eredetiből archiválva]. (Hozzáférés: 2008. március 15.)
- ↑ For example, Nature via Nurture: Genes, Experience and What Makes Us Human. Fourth Estate, 73. o. (2003). ISBN 978-1-84115-745-0
- ↑ (1964) „The Genain Quadruplets: A Case Study and Theoretical Analysis of Heredity and Environment in Schizophrenia”. Behavioral Science 9 (4), 371. o. DOI:10.1002/bs.3830090407.
- ↑ (2002. február 1.) „Signal transduction and the control of gene expression”. Science 295 (5556), 813–818. o. DOI:10.1126/science.1066355. PMID 11823631.
- ↑ Alberts et al. (2002), II.3. Control of Gene Expression – The Tryptophan Repressor is a Simple Switch That Turns Genes On and Off in Bacteria Archiválva 2007. június 29-i dátummal a Wayback Machine-ben.
- ↑ (2003. március 1.) „Epigenetic regulation of gene expression: how the genome integrates intrinsic and environmental signals”. Nature Genetics 33 (Suppl), 245–254. o. DOI:10.1038/ng1089. PMID 12610534.
- ↑ (2007. február 1.) „Paramutation: from maize to mice”. Cell 128 (4), 641–645. o. DOI:10.1016/j.cell.2007.02.007. PMID 17320501.
- ↑ Spontaneous mutations, An Introduction to Genetic Analysis, 7th, New York: W.H. Freeman (2000). ISBN 978-0-7167-3520-5
- ↑ (2004. április 1.) „Lesion (in)tolerance reveals insights into DNA replication fidelity”. The EMBO Journal 23 (7), 1494–1505. o. DOI:10.1038/sj.emboj.7600158. PMID 15057282.
- ↑ Induced mutations, An Introduction to Genetic Analysis, 7th, New York: W. H. Freeman (2000). ISBN 978-0-7167-3520-5
- ↑ (2013. február 1.) „DNA base damage by reactive oxygen species, oxidizing agents, and UV radiation”. Cold Spring Harbor Perspectives in Biology 5 (2), a012559. o. DOI:10.1101/cshperspect.a012559. PMID 23378590.
- ↑ (2012. július 1.) „DNA damage by reactive species: Mechanisms, mutation and repair”. Journal of Biosciences 37 (3), 503–517. o. DOI:10.1007/s12038-012-9218-2. PMID 22750987.
- ↑ Chromosome Mutation I: Changes in Chromosome Structure: Introduction, An Introduction to Genetic Analysis, 7th, New York: W.H. Freeman (2000). ISBN 978-0-7167-3520-5
- ↑ Encyclopedia of Microbiology. Academic Press, 551. o. (2009). ISBN 978-0-12-373944-5
- ↑ Environmental Biology. Cambridge University Press, 118. o. (2009). ISBN 978-0-521-67982-4
- ↑ (2007. április 1.) „Prevalence of positive selection among nearly neutral amino acid replacements in Drosophila”. Proceedings of the National Academy of Sciences of the United States of America 104 (16), 6504–6510. o. DOI:10.1073/pnas.0701572104. PMID 17409186.
- ↑ Variation and its modulation, An Introduction to Genetic Analysis, 7th, New York: W.H. Freeman (2000). ISBN 978-0-7167-3520-5
- ↑ Selection, An Introduction to Genetic Analysis, 7th, New York: W. H. Freeman (2000). ISBN 978-0-7167-3520-5
- ↑ (2001. november 1.) „Is the population size of a species relevant to its evolution?”. Evolution; International Journal of Organic Evolution 55 (11), 2161–2169. o. DOI:10.1111/j.0014-3820.2001.tb00732.x. PMID 11794777.
- ↑ Random events, An Introduction to Genetic Analysis, 7th, New York: W.H. Freeman (2000). ISBN 978-0-7167-3520-5
- ↑ On the Origin of Species. John Murray, 1. o. (1859). ISBN 978-0-8014-1319-3
Earlier related ideas were acknowledged in On the Origin of Species, 3rd, John Murray, xiii. o. (1861). ISBN 978-0-8014-1319-3 - ↑ (2003. október 1.) „Perspective: models of speciation: what have we learned in 40 years?”. Evolution; International Journal of Organic Evolution 57 (10), 2197–2215. o. DOI:10.1554/02-727. PMID 14628909.
- ↑ (2002. szeptember 1.) „Genome trees and the tree of life”. Trends in Genetics 18 (9), 472–479. o. DOI:10.1016/S0168-9525(02)02744-0. PMID 12175808.
- ↑ The Use of Model Organisms in Instruction. University of Wisconsin: Wisconsin Outreach Research Modules. [2008. március 13-i dátummal az eredetiből archiválva]. (Hozzáférés: 2008. március 15.)
- ↑ NCBI: Genes and Disease. NIH: National Center for Biotechnology Information. [2007. február 20-i dátummal az eredetiből archiválva]. (Hozzáférés: 2008. március 15.)
- ↑ (2003. február 1.) „'Mendelian randomization': can genetic epidemiology contribute to understanding environmental determinants of disease?”. International Journal of Epidemiology 32 (1), 1–22. o. DOI:10.1093/ije/dyg070. PMID 12689998.
- ↑ Pharmacogenetics Fact Sheet. NIH: National Institute of General Medical Sciences. [2008. május 12-i dátummal az eredetiből archiválva]. (Hozzáférés: 2008. március 15.)
- ↑ (2004. október 1.) „Genetic predisposition to cancer - insights from population genetics”. Nature Reviews. Genetics 5 (10), 764–772. o. DOI:10.1038/nrg1450. PMID 15510167.
- ↑ Human Molecular Genetics 2, second, John Wiley & Sons Inc. (1999) Chapter 18: Cancer Genetics Archiválva 2005. szeptember 26-i dátummal a Wayback Machine-ben.
- ↑ Lodish et al. (2000), Chapter 7: 7.1. DNA Cloning with Plasmid Vectors Archiválva 2009. május 27-i dátummal a Wayback Machine-ben.
- ↑ (2015. július 1.) „Artificial cloning of domestic animals”. Proceedings of the National Academy of Sciences of the United States of America 112 (29), 8874–8878. o. DOI:10.1073/pnas.1501718112. PMID 26195770.
- ↑ Lodish et al. (2000), Chapter 7: 7.7. Polymerase Chain Reaction: An Alternative to Cloning
- ↑ Section 2, Chapter 6: 6.1. The Methodology for DNA Sequencing, Genomes 2, 2nd, Oxford: Bios (2002). ISBN 978-1-85996-228-2
- ↑ Brown (2002), Section 2, Chapter 6: 6.2. Assembly of a Contiguous DNA Sequence Archiválva 2007. február 8-i dátummal a Wayback Machine-ben.
- ↑ (2006. március 1.) „Gene sequencing. The race for the $1000 genome”. Science 311 (5767), 1544–1546. o. DOI:10.1126/science.311.5767.1544. PMID 16543431.
- ↑ (2007. május 1.) „Advanced sequencing technologies and their wider impact in microbiology”. The Journal of Experimental Biology 210 (Pt 9), 1518–1525. o. DOI:10.1242/jeb.001370. PMID 17449817.
- ↑ (2006. január 1.) „Genomes for all”. Scientific American 294 (1), 46–54. o. DOI:10.1038/scientificamerican0106-46. PMID 16468433. Sablon:Subscription required
- ↑ „Scientists Seek Ban on Method of Editing the Human Genome”, The New York Times, 2015. március 19.. [2015. március 19-i dátummal az eredetiből archiválva] (Hozzáférés: 2015. március 20.)
- ↑ „A Powerful New Way to Edit DNA”, The New York Times, 2015. március 3.. [2015. március 26-i dátummal az eredetiből archiválva] (Hozzáférés: 2015. március 20.)
- ↑ (2015. április 1.) „Biotechnology. A prudent path forward for genomic engineering and germline gene modification”. Science 348 (6230), 36–38. o. DOI:10.1126/science.aab1028. PMID 25791083.
- ↑ (2015. március 1.) „Don't edit the human germ line”. Nature 519 (7544), 410–411. o. DOI:10.1038/519410a. PMID 25810189.
- ↑ „Chinese Scientists Edit Genes of Human Embryos, Raising Concerns”, The New York Times, 2015. április 23.. [2015. április 24-i dátummal az eredetiből archiválva] (Hozzáférés: 2015. április 24.)
- ↑ (2015. május 1.) „CRISPR/Cas9-mediated gene editing in human tripronuclear zygotes”. Protein & Cell 6 (5), 363–372. o. DOI:10.1007/s13238-015-0153-5. PMID 25894090.
Further reading[szerkesztés]
- Essential Cell Biology, 4th Edition. Garland Science (2013). ISBN 978-1-317-80627-1
- An Introduction to Genetic Analysis, 7th, New York: W. H. Freeman (2000). ISBN 978-0-7167-3520-5
- Genetics: Analysis of Genes and Genomes, 6th, Jones & Bartlett (2005). ISBN 978-0-7637-1511-3
- A Dictionary of Genetics, 8th, New York: Oxford University Press (2013). ISBN 978-0-19-976644-4
- Molecular Cell Biology, 4th, New York: Scientific American Books (2000). ISBN 978-0-7167-3136-8
External links[szerkesztés]
- Sablon:Wikiquote-inline
- Sablon:Wikibooks-inline
- Sablon:Library resources about
- Sablon:In Our Time
- Sablon:Curlie
Sablon:Genetics Sablon:Chromosome genetics Sablon:Branches of biology Sablon:Biology nav

